DNAシーケンサー373A(Strech型)
Key Word: DNAポリメラーゼ/蛍光検出/画像解析/クロマトグラム/塩基配列
はじめに
DNAの塩基配列決定技術は、遺伝子の構造機能を研究することはもとより進化系統の研究や個体識別鑑定を始め様々な目的に用いられる汎用技術となっている。特に、蛍光色素を用いて検出と配列分析が自動化されたオートマチックDNAシーケンサーが開発市販されて以来、塩基配列決定が著しく効率化されたこともこの技術が広く普及した要因となっている。本章では、理学部で共同利用されているDNAシーケンサー373A(Strech型)を中心に原理と使用方法等について解説する。
目次
______________________________
開発の背景
原理と性能
試料作成の材料の準備
試料作成の実際
ゲル作成の要点
機械の操作
データの修正と統合化
引用文献
______________________________
塩基配列の決定方法が分子生物学の研究に一般化したのはMaxamとGilbertにより化学的限定分解方法(Maxam&Gilbert法/1977年、文献1)が確立されて以来である。Sanger等によるDNAポリメラーゼとダイデオキシヌクレオチドを用いる方法(チェーンターミネーション法あるいはダイデオキシ法/1977年、文献2)も同時期に開発された。Maxam、GilbertとSangerはこの功績によりノーベル化学賞を授与されている。Maxam&Gilbert法は論文発表以前に研究者の間に広まり、この前後数年間世界中で利用された。その後はダイデオキシヌクレオチド等の試薬が市販されるようになってから、チェーンターミネーション法による決定方法が主流となり、現在に至っている。
チェーンターミネーション法は完成された手法であるものの、アイソトープを大量に用い、反応/電気泳動分離/検出/配列の読みとりまで全て研究者による手作業で時間と労力を多く必要とする。また、アイソトープを用いるため専用施設を必要とし、実験操作中の被爆/汚染を常に考慮して実験する必要があった。1980年代にアイソトープに代わって蛍光色素を用いる方法が考案されて、さらに検出を自動的に行う機械(DNAシーケンサー)が1986年に発表された。それ以来、蛍光物質/検出方式/酵素の改良などが各社で競合して行われた。現在ではアイソトープ法よりも反応や器具操作の面でも簡便で精度も高くなったため、検出や配列読み取りの自動化のメリットと相まって、広くDNAシーケンサーが利用されている。
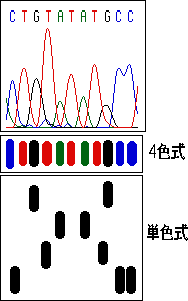
4種類の蛍光物質を同時に用いる方式(4色式、文献3)をもつPerkinElmer-ABI社の機械と1種類の蛍光物質のみを用いる方式(単色式、文献4)のAmersham-Pharmacia社、日立、島津製作所、Aloka社の製品がある。反応試薬の蛍光標識位置で分類するとオリゴヌクレオチド(プライマー)に蛍光色素を共有結合したものを用いる方式(ダイプライマー法)とダイデオキシヌクレオチド(ターミネーター)に蛍光色素を共有結合したものを用いる方式(ダイターミネーター法)がある。各社の製品ともこの2方式に対応可能となっている。
1種類の蛍光物質のみを用いる場合(単色式)は、アイソトープを用いたダイデオキシ法の場合と同様に1つのサンプルDNAについて4種の反応を別々に行い、4種の反応産物をそれぞれ別個に電気泳動分離検出する。4レーンのデータを1つにまとめて1つのサンプルDNAの塩基配列データになる。試薬、検出機械機構が単純化でき、試薬も安価ですむ。一方、4種類の蛍光物質用いる場合(4色式)は、試薬・装置とも高価であるが、単色式に比べて反応操作が少なくてすむことと、電気泳動で1サンプル当たり1レーンしか占拠しないので多数サンプルを同時に扱える長所がある。
共同利用のシーケンサーはアクリルアミドゲルによる分離方式で4色蛍光試薬を用い最大36個のサンプルを同時に扱うことができる。蛍光検出はレーザー励起の色フィルター分解(バンドパスフィルター)式となっている。Strechタイプに改変されているため700-800塩基長までの分離能を持っており、分離能では最新のシーケンサーと同等となっている。
試料作成に利用される試薬はキット化されて市販されているので、ここでは実験者自身で用意しなければならないDNA試料とプライマーについて説明する。
DNA試料
DNAシーケンサーの試料作成に用いられるテンプレートDNAの量
| DNAの種類 | サイクルシーケンス反応 | T7 polymerase反応 | ssDNA | 50-100ng | 200ng | Plasmid | 300-500ng | 5000ng | Cosmid, lambda Phage | 1000-2000ng | PCR Frgt. | 30ng- |
プライマー
ユニバーサルプライマーの利用 pUC19などの汎用プラスミドではクローニングサイト近傍の配列を持つプライマーが市販されている。M13-21, T7, T3プライマーなどよく用いられる汎用プライマーはユニバーサルプライマーと呼ばれている。これらは汎用プラスミドにクローニングされたDNAの配列を読むのに利用されている。ユニバーサルプライマーに蛍光物質を付加したものも市販されているのでダイプライマー法の反応にも利用できる。ユニバーサルプライマーを用いる場合にはクローン化された DNA断片の両端からそれぞれ500塩基程度しか配列を読むことができない。ユニバーサルプライマーを利用してより内部の塩基配列を分析するためには、DNA断片の片端から少しずつ異なった長さに欠失を作成したプラスミドDNA試料を作る必要がある(サブクローニング法、ネステドデリーション法など)。
カスタムプライマーの利用 塩基配列を読みたい部位の50-200塩基上流域の配列を化学合成して利用する。G+C含量が50%程度の場合16塩基長あればアニーリング温度50度の反応に利用するのに十分である。最低限に必要なプライマーの要件は融解温度の他に試料中にユニークな配列であることである。プライマー内に相補的な配列がない、著しい塩基の偏りがないこと等も望まれるが、必ずしも全ての要件を満たす配列を用いることができない場合に筆者等は3’端の近傍で4種の塩基がまんべんなく分布しているもので良い結果を得ている。プライマーデザインのためのソフトも市販されているので利用されたい。化学合成は1塩基長あたり100-200円で業者に依頼できるが、合成後の精製度に応じて値段はさらに高くなる。DNA合成業者はシーケンス反応に利用するオリゴヌクレオチドとしてHPLC精製グレードを薦めるが、筆者等は脱塩グレードを用いて経費を節約し十分な結果を得ている。
ダイプライマーの合成および精製方法についてはPerkinElmer-ABI社のプロトコールを参照されたい。合成は高価で手間もかかるので、一般の利用は少ないと考えられるが、ユニバーサルプライマーを利用できない特殊なベクターを用いて多数のサンプルを解析するような場合には有効と思われる。
チェーンターミネーション法でターミネーターとして用いられるダイデオキシヌクレオチドはデオキシヌクレオチドと競合関係にあり、Taqポリメラーゼなどの耐熱性DNAポリメラーゼの反応に取り込まれにくい。蛍光色素を保持しているダイターミネーターは、ダイデオキシヌクレオチドに比べてさらに耐熱性DNAポリメラーゼの反応に取り込まれにくいため、サイクルシーケンス反応はDNAシーケンサー用の試料作成法としては普及しなかった。しかし、耐熱性DNAポリメラーゼの改良が行われてダイターミネーターの取り込み効率が向上した結果、現在では最も操作が単純で少量の試料で着実な結果を出せる反応法となっている。
PerkinElmer-ABI社のキットを用いる場合の反応例を以下に記載する。
((*)ABI PRISM Dye Terminator Cycle Sequencing Ready Reaction Kit)
操作が簡単で、試料DNA量も少なくてすむことが解る。
_______________________________________________________
| Terminator Ready Reaction Mix(*) | 8microL | Primer(3microM) | 1microL | Plasmid 溶液(0.5microgram) | 11microL |
ゲル作成用の試薬はできるだけ高純度試薬を用いる。特にアクリルアミド溶液やウレアは古いものを用いない。アクリルアミドとウレアの溶液をイオン交換樹脂で処理することは筆者等は行っていないが、重合化触媒の添加の前に減圧脱気を兼ねてフィルター濾過をしている。
ゲルの固まるスピードも要因となる。ゲル溶液注入時に気泡ができると注入を中断しなければならなくなる。中断を考慮に入れて、気泡を追い出すためのゆとり時間を確保するため、ゲル溶液を冷却するという努力をする利用者もいる。気泡の発生を避けるためにガラス板は汚れや埃の無いようにし、取り扱いになれた人の操作を観察して修得する。また、ゲルの固化速度は室温やガラス板の温度に依存する。筆者等は重合化触媒のうち過硫酸アンモニウム液の量を夏期は半分に減らして固化スピードを調節している。注入後の固化時間は2時間程度で十分だと言われており、5時間ほどの間に泳動を開始している。
シグナルの検出部位はゲル下端から10センチ程度上の縦1センチの領域である。特にこの部分に傷や汚れがつかないようにガラス板を取り扱う。この部分周辺以外ではゲル板からウレアやゲル片をきれいに除去できていれば問題ない。ゲル固化後に装置本体へ装着する前のアルコールでの拭き取りに際しては、上記のレーザー照射部のみ簡単に行う。広い面積にわたりふき取ろうとすると、かえって汚れを広げて傷を多くつけることにもなるので、注意する必要がある。
機械の操作は設置されている先端機器の中では最も単純であり、既に操作になれた人と2回程度一緒に操作をすれば修得できる。以降はマニュアルを見ながら実行できるので活用されたい。
最も利用頻度の高い機械なので利用時間を守ることとトラブルが起きたときに速やかに対処する必要がある。夜に運転を開始してトラブルが起こった時、メーカーに連絡しても技術者は既に帰宅している。トラブルの対処は翌日以降になり、解決が長引く分だけ以降の利用予定者にしわ寄せされる。午後早くに運転を始めるのがトラブルを最小最短期間にするコツである。
機械の操作メニュー
1 機械の予約と運転に必要な時間
2 機械操作の実際
x 電気泳動を開始する時刻が遅れた場合
######################
1 機械の予約と運転に必要な時間
######################
DNAシーケンサーの先約状況の確認と利用予約はon lineでできる。
(cellvx.bio.sci.hiroshima-u.ac.jp)
利用予約をした日に利用する。
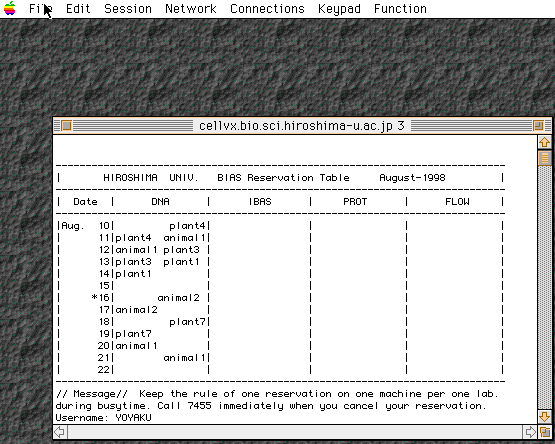
予約画面
本装置でロングゲルを用いると、起動してからデータの印刷までに約20時間かかる。
翌日の利用予約者が午後から利用できるように、機械利用は遅くとも午後5時には開始する。15時間のゲル電気泳動で800塩基長までのシグナル検出が行われる。メーカーのマニュアルでは18時間の泳動を薦めているが、15時間以上に長時間電気泳動しても、シグナルは得られるものの分離能の限界を超えているので実質的に無駄となることが多い。機械の利用を午後4時に始めれば終夜運転し翌日の朝8時頃に電気泳動が終了する。自動解析に1時間程度、印刷に1-2時間かかる。マニュアルレーントラッキングを朝9時から始め20分後に解析と印刷を始めると、午後1時には全ての操作が終了する。
機械の操作メニューへ戻る
メニューへ戻る
######################
2 操作の実際
######################
コンピューターの初期画面
装置本体とコンピュータの電源を入れる。

コンピュータでcellvxのアイコンでtelnet接続し、ユーザー名+パスワードでログインする。
4.DNA にてDNAシーケンサーを利用開始することを連絡する。
確認を求めてくるので、4+リターンする。
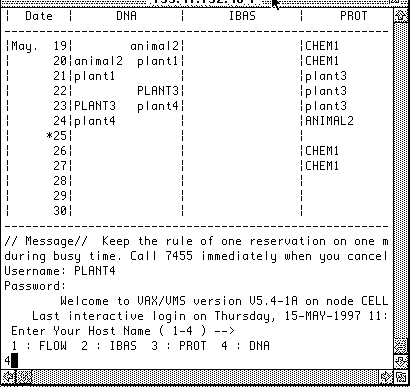
利用開始宣言
課金開始の表示があり、自動的にログオフされる。
メニューバー内の終了を選びtelnetプログラムを終了する。
Data Collectionのアイコンで機械本体からのデータ受信可能な状態にする。
以上の操作の間に装置本体はセルフチェックを終えてディスプレーにMain Menuが表示される。
電気泳動条件を設定する。(ロングゲルの場合は2800V 40mA 42W 15Hr)
Main Menuに戻り、PreRunメニューに移る。
ゲル板のレーザー照射される部分をイソプロパノールを少量つけた紙で軽く拭く。
固化したゲル板は次の作業を済ませておく。テープをはずして水道水で表面をよく洗いウレアやゲル片を洗い落としておく。蒸留水かイオン交換水でリンス後に乾かす。
本体の泳動部扉を開き下部バッファー槽とゲル板を装着し固定する。泳動部扉を閉じる。
PreRunメニューでPlate Checkを開始する。
ゲルに蛍光検出の障害となるような問題がないか検査するのがPlate Checkである。
コンピュータ画面にゲル板の走査状況が表示される。走査線に乱れがあればゲル板に汚れや傷があるので、装置から取り外して表面を確認し汚れを取る。汚れや傷が表面でなく内部の場合は取り除けないので、その位置に相当するレーンにサンプルを載せないよう位置を確認しておく。位置は画面上で走査線の乱れた位置にマウスのポイントを置くとその座標が表示されるので確認できる。
上部バッファー槽とサンプルコームを装着し、TBEバッファーを加える。
漏電やショートなど甚大なトラブルの元になるので、バッファーが漏れたら速やかにふき取る。バッファー注入後に上部泳動槽とゲル板との間から漏れがないかもチェックする。
泳動部扉を閉じてPreRunメニューでPreRunを開始する。
本体の表示部に実電圧と電流が表示されるので必ず数値を確認する。
ここで1000V以上加圧されていなければ、操作を中断する。
ゲルおよびバッファーもしくは機械のトラブルが想定される。
PreRun時点での確認でトラブルを知れば、試料以前の問題を未然に回避でき、高価な試料を無駄にしないですむ。

PreRun後に、サンプルウェルをリンスしてからサンプルを注入する。
リンスが不十分だとサンプルが乱れてシグナルがシャープでなくなり分離能が下がる。
サンプル数が多い場合は奇数レーンにアプライして10分間泳動し、再度リンスをして偶数レーンにアプライする。Main MenuからRunを指示して泳動を開始できる。
コンピュータ側では、メニューバーからsettingを選ぶ。
データ取り込み時間やサンプル数、プライマーの種類、自動解析や印刷についての指示をする。
メニューバーからNew sample file作成にはいる。
サンプルファイルの名前やコメント、解析や印刷についての個別の指示がここでできる。
最後に、本体から送信されてくるシグナルをコンピューターがハードディスクに保存しているか確認して、ディスプレイを暗くし終夜運転する。保存を指示できていないと、いっさいのデータは失われる。
翌朝、本体のディスプレイではRun Completedと表示されている。
途中にトラブルがあればエラーメッセージがでるので必ずメモする。
装置本体の電源を切る。
バッファー・泳動槽とゲル板を取り外す。
上部のバッファーを汲み出して少量にしてから、ゲル板と上部バッファー槽が合体したまま本体から取り外すのが、バッファーを本体に漏らさないコツである。本体にバッファーが漏れていれば少量でも必ずふき取る。バッファーの漏れが検出部や機械電気部におよぶと最悪である。この影響は次回からの利用者に出現する。
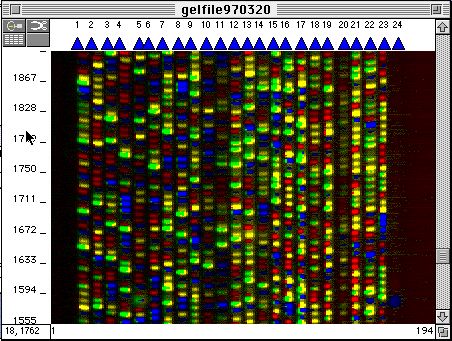 ゲルイメージ
ゲルイメージコンピュータ側では、転送されたシグナルをゲルファイルに貯めて、これを基にゲルイメージを作る。ゲルイメージ上のラダーをたどってレーントラッキングされる。サンプルファイルは自動的に作られる。自動解析を指示してあれば、Analysisソフトが起動されて各サンプルの解析が始まる。
自動レーントラッキングは必ずしも正しくない。特に、左側にサンプルが無い場合やシグナルの弱いサンプルが多いと、コンピュータはレーンを確実に見誤る。少なくとも、ゲルイメージの概観から各レーンに白線が誤り無い順番と位置で引かれているか確認する。
レーントラッキングを手動で行った場合、自動解析と印刷を指示する。解析データは先ず、Rawデータとなり、これを基にクロマトグラムと塩基配列が抽出される。いずれもサンプルファイルに書き込まれる。
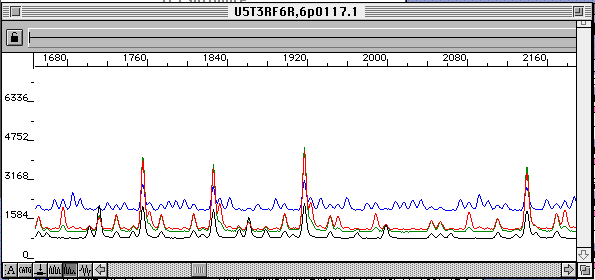
サンプルファイルとゲルイメージのデータ(ゲルファイル)を各自のメディアに保存する。共用コンピュータに長く貯めないように心がけたい。
###########################
x 電気泳動を開始する時刻が遅れた場合
###########################
電気泳動を開始するのが遅れた場合は、自動解析あるいは自動印刷の設定をしないでおく。電気泳動終了後にGelファイルのみ利用者のフォルダーへ移動しておけば、後日に解析や印刷から再開できる。Gelファイルを別のコンピュータで解析することも可能である。
メーカーのマニュアルでは18時間の泳動を薦めているが、15時間以上に長時間電気泳動しても、シグナルは得られるが分離能の限界を超えているので実質的に無駄となることが多い。15時間のゲル電気泳動で800塩基長程度までのシグナル検出が行われる。データの利用目的に応じて最小限の電気泳動時間に抑える。
自動解析や印刷の間に、ゲル板をはずし機械部分の拭き取りやベッファー槽の洗浄をすれば待ち時間を有効利用できる。
機械の操作メニューへ戻る
メニューへ戻る
#################
データの修正
#################
データのチェックと修正はクロマトグラムを印刷して行い、これを基に各自のコンピュータ上でEditViewプログラムを用いてデータファイルに修正を加える。
クロマトグラムのプライマー近傍ではアーテファクトのシグナルが多いので塩基を削除する。
また、600塩基を越えて、同じ塩基が連続した部分などでピークが分かれにくくなるので誤りが多い。以降を削除する。読みとり開始位置をずらして取った複数データを重ね合わせるために、多少ピークの分離の悪くなった部分を残しておくこともある。
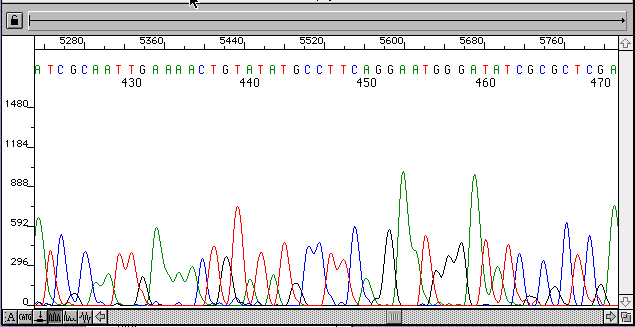
EditViewあるいはAnalysisプログラムでデータファイルに修正を加えると並列の塩基配列のみのテキストデータも修正される。
メニューへ戻る
#################
データの統合化
#################
________________________________________________________
1つの遺伝子やcDNAでも1サンプルの塩基配列データのみでは全配列を読むことはできないので、数個から数十個のデータをつなぎ合わせる必要がある。
相補鎖DNAの配列とつないだり対比させる必要もある。ここで用いるソフトウェアについてふれておく。
________________________________________________________
従来からある塩基配列解析ソフト、例えばGenetyx(ソフトウェア開発)では、塩基配列データ(テキスト)を基に配列の結合統合化を行う機能が備わっているものもあるので、配列データの統合編集に利用できる。
編集しようとする塩基配列間でデータに矛盾が生じることもしばしばある。生データに近い情報を基にどちらのデータが正しいのか判定する必要がでてくる。 大きい遺伝子やオペロンの分析(当然に高い精度で行われる)では配列決定に占める検証の割合が高くなり、データ数も多いので複雑である。これらの作業を効率的に行うためのソフトウェアが開発されている。
SeqEdとAutoAssemblerはABI社が開発したプログラムで、同社のシーケンサーのデータを複数同時に表示対比可能でデータファイルに修正を加えることもできる。DNA sequencher(Gene Code Corp.)では、データファイルに修正を加えることはできないが,複数社のシーケンサーのデータを分析編集できる点で優れている。いずれも高価なソフトであるが、編集時の見落としやエラーは塩基配列の精度に大きく影響するので導入を薦めたい。
1) Maxam A.M., Gilbert W. (1977) Proc. Natl. Acad. Sci. U.S.A. 74:560-564.
2) Sanger F., Nicklen S., Coulson A.R. (1977) Proc. Natl. Acad. Sci. U.S.A. 74:5463-5467.
3) Smith L.M., Sanders J.Z., Kaiser R.J., Hughes P., Dodd C., Connell C.R., Heiner C., Kent S.B.H., Hood L.E. (1986) Nature 321:674-679.
4) Ansorge W., Sproat B.S., Stegemann J., Schwager C. (1986) J. Biochem. Biophys. Methods 13:315-323.